В последние годы перспективным подходом в современной комбинированной химиотерапии онкологических заболеваний является направленная коррекция профиля экспрессируемых генов на постгеномном уровне с помощью малых молекул. Основой такого подхода является предпочтительное связывание аффинных к ДНК соединений разной структуры, как интеркаляторов, так и узкобороздочных лигандов, с определенными контекстами последовательности ДНК, которые существенны для транскрипции генов и функционирования белков «домашнего хозяйства». Последние исследования показывают, что эпигенетические способы воздействия на генотип клетки играют важную роль в канцерогенезе и прогрессии опухоли. При этом возникает вопрос о возможности обратной регуляции этих процессов с помощью различных ксенобиотиков [14], среди которых особый интерес представляют узкобороздочные лиганды (УБЛ) [4]. Данные соединения интересны как наиболее мягко действующие агенты, обладающие специфичностью к определенным последовательностям ДНК и способные менять характер взаимодействия белков «домашнего хозяйства» клетки с ДНК, в том числе влияя на уровни экспрессии генов путем изменения профиля метилирования ДНК и модификации гистонов [1].
Основными методами изучения взаимодействий ДНК-связывающих молекул с ДНК являются поверхностный плазмонный резонанс [16], ЯМР [17], рентгеноструктурный анализ [7] и футпринтинг [9]. Последний является наиболее простым и доступным методом, тем не менее позволяющим получать данные об изменении геометрии дуплексной структуры в местах связывания УБЛ. Изначально футпринтинг использовали для выявления специфических участков ДНК, распознаваемых различными белками, участвующими в метаболизме и функционировании ДНК [9]. В дальнейшем этот метод стали успешно применять для более тонкого изучения структуры ДНК-белковых комплексов [20], а также их кинетических и термодинамических свойств [13]. В основе метода лежит следующий принцип. Изучаемый белок, связываясь со специфической последовательностью ДНК-дуплекса, защищает ее от гидролизующего ДНК реагента, в то время как свободные от взаимодействия участки дуплекса подвергаются расщеплению. Участок связывания белка определяют по последовательности, не подвергнувшейся расщеплению. Продукты расщепления визуализируются с помощью электрофореза в ПААГ в денатурирующих условиях. О связывании белка с некоторой областью ДНК свидетельствует уменьшение интенсивности зон на электрофореграмме, соответствующих продуктам расщепления ДНК в этой области. Обычно для визуализации ДНК метят по одному из 5’-концов фосфатом, содержащим изотоп 32P. Возможность регистрации малых количеств изотопа позволяет использовать минимальное количество ДНК, что имеет существенное значение в связи с недоступностью многих ДНК-связывающих белков в больших количествах (в случае футпринтинга комплексов УБЛ c ДНК такая проблема отсутствует). Флуоресцентная метка используется для специальных методов футпринтинга [19, 10, 11].
Целью представленного исследования была оптимизация футпринтинга ДНКазой I путем использования флуоресцентно-меченных ДНК-дуплексов и проведение сравнительного анализа взаимодействия ряда УБЛ с ДНК.
Материалы и методы исследования
Узкобороздочные лиганды: Hoechst 33342 – 2’-(4-этоксифенил)-5-(4-метил-1-пиперазинил)-2,5’-бис-бензимидазола тригидрохлорид, Hoechst 33258 – 2’-(4-гидроксифенил)-5-(4-метил-1-пиперазинил)-2,5’-бис-бензимидазолтригидро-хлоридгидрат; DAPI – 4’,6-диамино-2-фенилиндол; Diminazene – 4,4’-(1-триазен–1,3–дийил)бис-бензенекарбокси-мидамид; Pentamidine – 4,4’-(пентаметилендиокси)дибензамидин. В качестве растворителей использовали диметилсульфоксид (DMSO) («ПанЭко», Россия) и воду, очищенную системой milliQ.
Флуоресцентно-меченные фрагменты ДНК были получены методом ПЦР-амплификации. В качестве матрицы были использованы плазмиды pRFPCer [12] и pUC19. Реакцию проводили на амплификаторе PTC-100 («MJ Research Inc», Канада) в 50 мкл реакционной смеси следующего состава: ПЦР-буфер («Синтол», Россия), dNTP (0,2 мМ), хлорид магния (2 мМ), Taq-полимераза (2,5 е.а.), 1 нг матрицы и по 10 пмоль каждого из праймеров (последовательность праймеров представлена в таблице). 30 циклов ПЦР проводили по следующей программе: 94 °С – 30 с, 56 °С – 60 с, 72 °С – 40 с. Фрагменты ДНК были очищены методом электрофореза в 10 %-ном ПААГ в неденатурирующих условиях в TBE-буфере с последующим выделением из геля 2 М водным раствором перхлората лития в течение 14 ч при комнатной температуре. ДНК осаждали ацетоном. Пробы центрифугировали, осадок промывали ацетоном и высушивали.
Флуоресцентно-меченные фрагменты ДНК, использованные в работе
|
Название |
Последовательность праймеров для ПЦР, 5’ → 3’ |
Плазмида |
|
d159(T/F/R/C3/C5) |
TGTGCTGCAAGGCGATTAAG; (FAM/ROX/TAMRA/Cy3/Cy5,5)TCGGGAAACAGCTATGACCATG |
pRFPCer |
|
d200T |
ctctagtgtcgacctgcaggcatg; (TAMRA)ATATTTTCCCGACTGGAAAGCGGG |
pUC19 |
Комплексообразование узкобороздочного лиганда с ДНК проводили в течение 15 минут при 37 °С в 20 мкл реакционной смеси следующего состава: буфер А (10 mM Tris-HCl (pH 7,5), 2,5 mM MgCl2, 1 mM CaCl2), флуоресцентно-меченные фрагменты ДНК (0,2 мкМ) и узкобороздочный лиганд (8 мкМ). Затем добавляли 3 мкл 0,5 мкЕА/мкл ДНКазы I (оптимальная концентрация фермента была подобрана в предварительных экспериментах), инкубировали 15 минут при 37 °С. Реакцию останавливали добавлением 15 мкл формамида и нагреванием до 95 °С в течение 5 мин.
Пробы анализировали в денатурирующих условиях в ПААГ (11,5 % акриламида, 0,5 % N,N’-метиленбисакриламида, 7 М мочевина и 20 % формамид) толщиной 4 мм в TBE-буфере при напряженности поля 8 В/см. Пробы наносили на гель без предварительной обработки. Ячейки геля промывали TBE-буфером дважды: перед нанесением проб, а также через 10–20 с после начала электрофореза (при напряженности электрического поля 2 В/см). Последняя процедура позволяет избавиться от нежелательных солей буфера, используемого в реакции связывания. ДНК, являясь полианионом, проникает в гель значительно быстрее, чем однозарядные анионы, поэтому в момент второго промывания ячеек ДНК уже находится в геле, в то время как соли удаляются, что значительно улучшает разделение продуктов гидролиза ДНК.
Визуализацию результатов и обсчет данных проводили на приборе Thyphoon 9410 («GE Healthcare», США), используя компьютерную программу ImageQuant TL 7.0. Гель сканировался без отделения от стекол. Параметры сканирования: Mode – Fluorescence, Pixel size-200 micron, Focal Plate + 3 mm, Sensitivity – Normal, PMT-800, Filter/Laser-526 SP/Green (532) для FAM, 580 BP/Green (532) для TAMRA и Cy3, 670 BP/Red (633) для Cy5,5, 610 BP/ Green (532) для ROX.
Результаты исследования и их обсуждение
В наших экспериментах были использованы известные УБЛ – Pentamidine, Diminazene, Hoechst 33258 и Hoechst 33342, DAPI. Выбор анализируемых соединений был обоснован их применением в медицине или в молекулярно-биологических исследованиях. В качестве основы разрабатываемой модификации метода футпринтинга были взяты протоколы процедур с использованием радиоактивно меченной ДНК [6, 5, 8].
Оптимизация условий футпринтинга
Футпринтинг ДНКазой I является основным методом изучения сиквенс-специфичности узкобороздочных лигандов [18, 15, 2]. Для футпринтинга ДНКазой I с использованием флуоресцентной метки, так же, как и в случае радиоактивной метки, нами были использованы фрагменты ДНК длиной от 150 до 200 п.о. Их последовательность выбиралась так, что предполагаемый сайт узнавания был расположен ближе к центру фрагмента. Нами была подобрана концентрация ДНКазы I для проведения реакции гидролиза этих фрагментов ДНК. Необходимым условием успешного проведения футпринтинга является такая активность ДНКазы I, при которой молекула ДНК будет гидролизована только по одному положению, при этом число гидролизованных молекул ДНК не должно превышать 30 % от их общего количества. Невыполнение этого условия приводит к нечеткому профилю расщепления ДНК и трудностям в интерпретации результата эксперимента. Степень расщепления фрагмента оценивали по отношению интенсивности зоны неразрезанной ДНК к общей интенсивности всех полос образца. На рис. 1, А приведены результаты гидролиза ДНКазой I 159-звенного ДНК-дуплекса (d159T), взятого в различных концентрациях. Полученный результат свидетельствует о том, что концентрация 0,5 мкЕА/мкл является наиболее предпочтительной (степень гидролиза составляет 20 %) для проведения футпринтинга в буфере А.
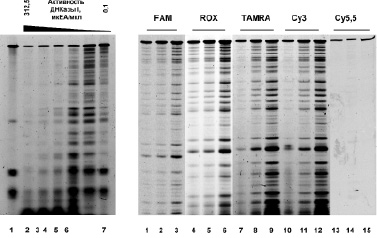
А Б
Рис. 1. Анализ продуктов гидролиза 159-звенных ПЦР фрагментов ДНКазой I: А – электрофореграммы расщепления 159-звенных ПЦР фрагментов (d159T), меченных флуорофором TAMRA, ДНКазой I, взятой в концентрациях 2–7: 312,5; 62,5; 12,5; 2,5; 0,5; 0,1 мкЕА/мкл. 1 – исходный 159-звенный ПЦР фрагмент; Б – электрофореграммы расщепления ДНКазой I, взятой в концентрации 0,5 мкЕА/мкл, 159-звенных ПЦР фрагментов (d159F), меченных флуорофорами FAM, ROX, TAMRA, Cy3 и Cy5,5, взятых в концентрациях 7, 10–0,05 мкМ; 1, 4, 8, 12–0,2 мкМ; 2, 5, 9, 12, 13–0,5 мкМ; 3, 14–1 мкМ; 15–2 мкМ
Для корректной оценки результатов футпринтинга использовали следующие контрольные образцы:
1) негидролизованная ДНК;
2) ДНК, гидролизованная в отсутствие ДНК-связывающей молекулы (контроль позволяет сравнивать интенсивность каждой из его зон с интенсивностью соответствующей зоны электрофореграммы расщепления комплекса;
3) анализ последовательности ДНК по Максаму – Гилберту (образец позволяет идентифицировать последовательность ДНК в участке связывания с молекулой).
Подбор флуоресцентной метки
В настоящее время известно много различных флуоресцентных красителей, обладающих различными свойствами. Выбор флуоресцентной метки обуславливается возможностью ее детекции, определяющей минимальное количество флуоресцентно-меченной ДНК, необходимое для визуализации результатов расщепления. С этой целью нами был протестирован ряд наиболее распространенных флуоресцентных красителей: FAM, Cy3, ROX, TAMRA, Cy5,5. Сканирование проводили на приборе Thyphoon 9410. На рис. 1, Б приведена электрофореграмма гидролиза 159-звенного ПЦР-фрагмента ДНКазой I, взятого в различных концентрациях, меченного выбранными флуорофорами. В соответствии с представленной электрофореграммой наибольшей чувствительностью обладают TAMRA и Cy3: минимальная концентрация, достаточная для визуализации ДНК в данных условиях, составляет 0,2 мкМ. Более высокая концентрация требуется для красителя ROX и составляет 0,3 мкМ. FAM и Cy5,5 видны при концентрациях 0,5 и 1 мкМ соответственно, что позволяет сделать вывод о нецелесообразности их использования в качестве флуоресцентных красителей для футпринтинга. Таким образом, TAMRA и Cy3 являются наиболее подходящими флуорофорами для футпринтинга (примечание: чувствительность ряда флуоресцентных сканеров, таких как Thyphoon 9500 (GE Healthcare», США) и FLA-3000 («Fujifilm», Япония), является не достаточной для нормальной визуализации расщепления ДНК).
Футпринтинг комплексов узкобороздочных лигандов с ДНК с использованием флуоресцентной метки
Ранее с помощью футпринтинга ДНКазой I в ряде работ была проанализирована сиквенс-специфичность отдельных УБЛ (Pentamidine, Diminazene, Hoechst 33258 и 33342, DAPI) и во всех этих исследованиях было показано, что данные УБЛ проявляют специфичность к AT-богатым участкам ДНК [13, 2, 13]. Комплексного сравнительного анализа взаимодействий нескольких УБЛ с ДНК ранее не проводилось. После оптимизации футпринтинга с использованием флуоресцентной метки нами был проведен сравнительный анализ «футпринтов» изучаемых УБЛ.
На рис. 2 приведены электрофореграммы расщепления 200-звенной ДНК, меченной флуорофором TAMRA, в присутствии DAPI, Hoechst 33349 и Hoechst 33258, Pentamidine, Diminazene, в 8 мкМ концентрации, а также в отсутствии этих соединений (дорожка 4). Для Hoechst 33349 и Hoechst 33258 наблюдается характерное изменение интенсивности зон в АТ-богатых областях – АТ-специфичность. Тем не менее, несмотря на близость химической структуры Hoechst 33349 и Hoechst 33258, существуют некоторые отличия в картине разрезания их комплексов с ДНК, что говорит о некоторых различиях во взаимодействии этих лигандов с ДНК. Похожую специфичность проявляет DAPI (дорожка 5), однако в этом случае «футпринт» не настолько выражен, как в случае соединений Hoechst, что свидетельствует о более низкой константе его связывания с ДНК. В целом это согласуется с литературными данными [2]. Среди изученных соединений наименьшее влияние на расщепление ДНК ДНКазой I оказывает Pentamidine, что объясняется тем, что это соединение не обладает ярко выраженной сиквенс-специфичностью.
Таким образом, в нашем исследовании для ряда УБЛ продемонстрированы тонкие различия в специфичности и способности к связыванию с ДНК, что является важной информацией при анализе эффектов УБЛ при их применении в комбинированной химиотерапии онкологических заболеваний. Полученные нами данные согласуются с ранее опубликованными результатами исследований, демонстрирующими AT-специфичность УБЛ.
Заключение
Нами был оптимизирован метод футпринтинга ДНКазой I с использованием флуорофора в качестве метки. Проведение исследований с использованием флуоресцентной метки вместо радиоактивной представляется значительно более безопасным. Флуоресцентная метка вводится либо непосредственно в изучаемый фрагмент ДНК в процессе автоматического синтеза, либо методом ПЦР с праймерами, имеющими флуорофор в своем составе.
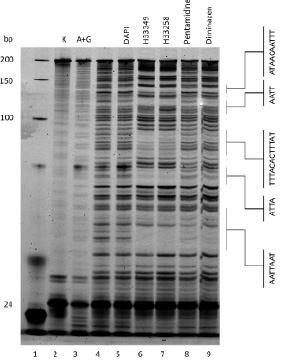
Рис. 2. Футпринтинг ДНКазой I флуоресцентно-меченных 200-звенных ПЦР-фрагментов (d200T) в присутствии 8 мкМ различных узкобороздочных лигандов: 1 – 24, 100, 150, 200-звенные ДНК; 2 – исходный 200-звенный ПЦР фрагмент; 3 – химическое расщепление по пуринам пиперазином; 4 – расщепление d200T ДНКазой I в отсутствие узкобороздочных лигандов; 5–9 – расщепление d200T в присутствии DAPI, Hoechst 33349, Hoechst 33258, Pentamidine, Diminazen
Использование флуорофоров значительно повышает экспрессность футпринтинга по сравнению с футпринтингом с использованием радиоактивной метки.
В результате работы показано, что наиболее предпочтительными флуоресцентными красителями являются TAMRA и Cy3. Их применение позволяет использовать минимальную концентрацию ДНК – 0,2 мкМ. Продемонстрировано, что футпринтинг ДНКазой I с использованием флуоресцентной метки представляет собой эффективный метод для определения предпочтительных участков узнавания ДНК-связывающих молекул, так же, как и применяемая ранее менее безопасная и более трудоемкая модификация метода, с использованием в качестве метки 32P. Кроме того, флуорофор позволяет визуализировать непосредственно электрофореграмму расщепления ДНК; а не его «отображение» на чувствительной к радиоактивному излучению пленки, как в случае использования радиоактивной метки. Показано, что такие близкие по структуре вещества, как Hoechst 33349 и Hoechst 33258, по-разному влияют на расщепление ДНК ДНКазой I, что согласуется с их биологическим действием на метилирование ДНК [1]. Таким образом, футпринтинг ДНКазой, с использованием флуоресцентной метки, позволяет получить данные о взаимодействии УБЛ с ДНК, объясняющие их биологические свойства. Ограничением метода является необходимость использования высокочувствительных флуоресцентных сканеров.
Исследование проведено при поддержке РФФИ, грант № 15-04-09216А.
Рецензенты:
Кубарева Е.А., д.х.н., профессор, главный научный сотрудник, Научно-исследовательский институт физико-химической биологии имени А.Н. Белозерского, ФГБОУ ВО «Московский государственный университет имени М.В. Ломоносова», г. Москва;
Бухман В.М., д.м.н., профессор, руководитель лаборатории фармакологии и токсикологии НИИ экспериментальной диагностики и терапии опухолей, ФГБНУ «Российский онкологический научный центр имени Н.Н. Блохина», г. Москва.



