Микробиологический мониторинг возбудителя туберкулеза в современных условиях основан на анализе специфических нуклеотидных последовательностей хромосомной ДНК M. tuberculosis с использованием методов, основанных на ПЦР – MIRU-VNTR-типировании и сполиготипировании, также IS6110-RFLP и других методов исследования геномного полиморфизма микобактерий [4–7]. Соотношение отдельных генотипов в популяциях M. tuberculosis может существенно различаться в разных странах и географических регионах мира, что следует учитывать при разработке стратегий генотипирования микроорганизма [7–10].
Цель данной работы – изучить региональные особенности генетической структуры популяции M. tuberculosis с помощью стандартизованных молекулярно-генетических методов и разработать схему генотипирования возбудителя туберкулеза в Уральском федеральном округе РФ.
Материалы и методы исследования
178 культур M. tuberculosis и 90 образцов ДНК, выделенных из клинического материала, были получены от вновь выявленных эпидемиологически не связанных больных туберкулезом в 2009–2012 гг. в лабораториях ГБУЗ СО «ПТД», г. Екатеринбург, и ФГБУ «УНИИФ» Минздрава России.
Культивирование M. tuberculosis осуществляли общепринятым методом на среде Левенштейна – Йенсена. Колонии M. tuberculosis суспендировали в растворе, содержащем 9 % NaCl и 20 % глицерина. Часть суспензии сохраняли в пробирках для последующего IS6110-RFLP-типирования, другие инкубировали при 95 °С – 30 мин для лизирования клеток, затем осаждали при 3000 g, 1 мин. Супернатант использовали в качестве препарата ДНК для постановки ПЦР.
Для дифференциации изолятов на группы Beijing и non-Beijing использовали ПЦР-тест-систему «Амплитуб-Beijing», ООО «Синтол», г. Москва. ПЦР выполняли в режиме реального времени на приборе iCiclerQ5 (BioRad, США).
Генотипирование изолятов M. tuberculosis проводили методом MIRU-VNTR, используя 15 локусов [5] и дополнительно три гипервариабельных локуса [10] для изолятов генотипа Beijing, сгруппированных в кластеры. Для постановки ПЦР использовали реактивы ООО «Интерлабсервис», г. Москва, и праймеры [2, 7] ООО «Синтол», г. Москва. ПЦР осуществляли в термоциклере «Терцик», ООО «ДНК технология». Продукты ПЦР подвергали электрофорезу в 2 % агарозном геле, окрашенном бромистым этидием, результаты визуализировали с помощью системы документации гелей «GelDoc», BioRad, США.
Сполиготипирование [6] и IS6110-RFLP-типирование [4] проводили на базе лаборатории молекулярной микробиологии ФБУН НИИ эпидемиологии и микробиологии имени Пастера, г. Санкт-Петербург. Принадлежность к генетической группе/линии (lineage, clade) определяли путем сравнения полученных профилей изолятов с имеющимися в компьютерных базах данных: «MIRU-VNTRplus» [1], в международной базе данных SITVITWEB [2] и ее обновленной версии SITVIT2. Профили IS6110-RFLP сравнивали с представленными в базе данных ФБУН НИИ эпидемиологии и микробиологии имени Пастера, г. Санкт-Петербург. Для количественной оценки вариабельности генетических локусов и дискриминирующей способности схем генотипирования рассчитывали индекс разнообразия Хантера - Гастона (HGDI) согласно [3].
Результаты исследования и их обсуждение
С помощью ПЦР в режиме реального времени определена принадлежность 178 изолятов M. tuberculosis и 75 образцов клинического материала, содержащих ДНК M. tuberculosis, к генетическим группам Beijing и non-Beijing.
Анализ результатов MIRU-VNTR-типирования (15 локусов) 178 изолятов M. tuberculosis выявил 107 типов (MIT) MIRU-VNTR-профилей.
В пределах генетической группы non-Beijing (n = 80) было выделено 64 MIRU-VNTR-профиля, которые представляли пять генетических групп: LAM (28,8 %), URAL (26,3 %), Haarlem (17,5 %), Tur, S и одну неклассифицированную группу (Unknown). Двадцать восемь изолятов (35 %) были сгруппированы в 12 кластеров, содержавших по 2–4 изолята в каждом. Анализ структуры DR-области хромосомы всех изолятов non-Beijing методом сполиготипирования позволил выделить 43 сполиготипа (SIT) 15 генетических семейств: наиболее многочисленное – Н3 (25 %) включало изоляты MIRU-VNTR группы URAL. Значения HGDI для MIRU-VNTR-типирования и сполиготипирования были достаточно высокими и составили 0,994 и 0,970 соответственно.
MIRU-VNTR-типирование (15 локусов) выявило неоднородность изолятов группы Beijing (n = 98), причем 65 (66,3 %) из них входили в состав 10 кластеров. Самый многочисленный из кластеров включал 32 (32,7 %) изолята (рис. 1). Данные генотипирования по трем дополнительным гипервариабельным локусам [10] были получены для 53 из 65 изолятов, что позволило разбить 7 из 10 MIRU-VNTR-кластеров Beijing.
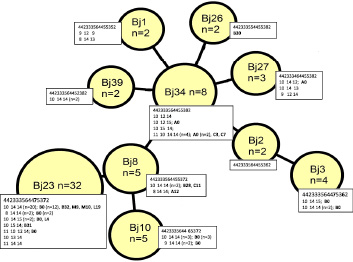
Рис. 1. Минимальное охватывающее древо (Minimum spanning tree) профилей MIRU-VNTR (15 локусов) клинических изолятов M. tuberculosis Beijing вновь выявленных больных туберкулезом
Размеры узлов пропорциональны числу изолятов в составе MIRU-VNTR-кластера. Числовой профиль (число повторов в каждом из 15 локусов – Mtub04, ETRC, MIRU04, MIRU40, MIRU10, MIRU16, Mtub21, QUB11b, ETRA, Mtub30, MIRU26, MIRU31, Mtub39, QUB26, QUB4156) показан в 1-й строке у каждого узла; ниже построчно приведены числовые профили для трех гипервариабельных локусов (VNTR4120, VNTR3820, VNTR3232) и профили IS6110-RFLP, обозначены жирным шрифтом. Цифры в скобках обозначают число изолятов, превышающее 1.
Самые большие значения HGDI (0,651 и 0,603) имели локусы MIRU26 и QUB26 соответственно. Локусы MIRU31, Mtub21, QUB11b, MIRU40 имели невысокую степень полиморфизма, значения HGDI: 0,243, 0,190, 0,173 и 0,155 соответственно. Для остальных локусов HGDI не превышал 0,100. Таким образом, был определен минимальный набор из 6 (MIRU26 QUB26 MIRU31, Mtub21, QUB11b, MIRU40) дискриминирующих локусов MIRU-VNTR для генотипирования изолятов Beijing (HGDI = 0,875). Использование данного набора, включавшего 6 из 15 «классических» локусов [5], несколько снижало и без того относительно невысокий уровень дискриминации изолятов (HGDI = 0,885). Напротив, включение трех дополнительных локусов [10] позволило существенно увеличить дискриминирующую способность 6-локусной схемы типирования (HGDI = 0,939).
Рекомендуемая нами схема MIRU-VNTR-типирования представлена на рис. 2.
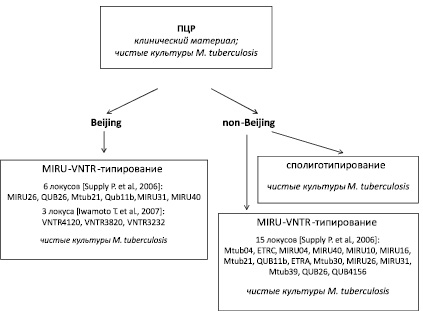
Рис. 2. Схема генотипирования изолятов M. tuberculosis
Данные IS6110-RFLP-типирования, полученные для 45 изолятов, еще больше повысили степень дифференциации в пределах MIRU-VNTR-кластеров (рис. 1). У изолятов M. tuberculosis Beijing выявлено 17 близкородственных (коэффициент сходства 83 %) типов IS6110-RFLP-профилей, которые различались как по количеству (15–19), так и по молекулярной массе фрагментов рестрикции хромосомной ДНК, содержащих участок последовательности инсерционного элемента IS6110. Пятнадцать (88 %) из 17 вариантов профилей рестрикции IS6110 были индивидуальны (т.е. обнаружены у одного изолята), остальные представляли два крупных кластера: А0 и В0, включавших 5 и 25 изолятов с идентичными профилями рестрикции соответственно. IS6110-RFLP-типирование, повысившее эффективность дифференциации изолятов M. tuberculosis, не может быть рекомендовано для широкого применения, поскольку является дорогостоящим и трудоемким методом, доступным лишь специализированным лабораториям.
Таким образом, для практического применения можно предложить схему генотипирования уральских изолятов M. tuberculosis, включающую на первом этапе их дифференциацию на группы Beijing и non-Beijing, с использованием ПЦР в режиме реального времени. Последующее генотипирование изолятов non-Beijing рекомендуется проводить с использованием 15 MIRU-VNTR-локусов [5] и / или сполиготипированием. Минимальный набор из 6 локусов стандартной схемы (MIRU26, QUB26, Mtub21, Qub11b, MIRU31, MIRU40) и 3 дополнительных локуса (VNTR4120, VNTR3820, VNTR3232) следует использовать для генотипирования изолятов генотипа Beijing (рис. 2).
Рецензенты:
Мордовской Г.Г., д.м.н., заведующий отделом лабораторной диагностики, Свердловский областной противотуберкулезный диспансер, г. Екатеринбург;
Чугаев Ю.П., д.м.н., профессор кафедры фтизиатрии и пульмонологии, Уральский государственный медицинский университет, г. Екатеринбург.
Работа поступила в редакцию 15.09.2014.
Библиографическая ссылка
Скорняков С.Н., Умпелева Т.В., Вязовая А.А., Кравченко М.А., Еремеева Н.И., Нарвская О.В. ГЕНОТИПИРОВАНИЕ УРАЛЬСКИХ ИЗОЛЯТОВ MYCOBACTERIUM TUBERCULOSIS // Фундаментальные исследования. – 2014. – № 9-11. – С. 2485-2488;URL: https://fundamental-research.ru/ru/article/view?id=35385 (дата обращения: 16.04.2024).



