Изучение генетического разнообразия и внутривидовой дифференциации лесообразующих видов хвойных растений, в том числе и видов рода Larix, имеющих важное биосферное и ресурсное значение, является одной из важнейших задач популяционной биологии. Особую актуальность исследование этих вопросов приобрело в связи с проблемой сохранения генетических ресурсов хвойных видов растений. В последние десятилетия в результате увеличения антропогенного давления на природные растительные сообщества и экосистемы возникла серьезная угроза сокращения их генетического разнообразия. Только на основании точных сведений о генетической структуре популяций хвойных, уровня их генетической изменчивости и характере ее распределения в пределах ареалов видов может быть оценен генетический потенциал видов и разработан для каждого из них комплекс мероприятий, направленных на максимальное сохранение генетического разнообразия в процессе их использования и воспроизводства [1].
Успешность решения задач общей и частной популяционной генетики многих видов зависит от изученности различных элементов генома и их полиморфизма [13]. В последнее время для этих целей широко используются микросателлитные [11, 14] и межмикросателлитные [17] или ISSR-маркеры (Inter Simple Sequence Repeats). В качестве праймеров для ISSR-анализа полиморфизма ДНК в ПЦР используют короткие ди- и тринуклеотидные микросателлитные повторы. Такие праймеры позволяют амплифицировать фрагменты ДНК, которые находятся между двумя достаточно близко расположенными инвертированными микросателлитами (как правило, это уникальная ДНК). В результате амплифицируется большое число фрагментов, представленных на электрофореграмме дискретными полосами (ISSR-фингерпринтинг). Полученные паттерны ПЦР-продуктов видоспецифичны. В геномах растений количество микросателлитных повторов очень велико, что делает этот метод удобным для генетического анализа. Микросателлитные последовательности окружают многие гены и могут быть использованы как якорные последовательности к этим генам, ISSR-маркирование не требует предварительного знания нуклеотидной последовательности исследуемой ДНК [7]. Метод обладает хорошей воспроизводимостью и может быть с успехом использован для выявления межвидовой и внутривидовой генетической изменчивости, идентификации видов, популяций, линий, а в ряде случаев и для индивидуального генотипирования [6]. Эти маркеры нашли широкое применение в популяционно-генетических исследованиях различных объектов [2, 4, 5]. Однако, поскольку геномное распределение микросателлитов существенно отличается у разных таксонов [7], а возможные причины таких отличий до сих пор недостаточно изучены, перед использованием ISSR-маркеров для изучения генетической изменчивости и идентификации видов необходимо исследовать полиморфизм спектров получаемых фрагментов ДНК в зависимости от корового мотива микросателлита [12]. В связи с чем в данной работе выполнен сравнительный анализ полиморфизма ISSR-маркеров, полученных с использованием в качестве праймеров разных мотивов микросателлитов у популяций лиственницы различного происхождения.
Целью данного исследования являлся анализ полиморфизма ISSR-маркеров и генетического разнообразия природной и искусственной популяций лиственницы Пермского края.
Материал и методы исследования
Сбор растительного материала (хвои) произведен с 26 случайно выбранных деревьев искусственных насаждений лиственницы, расположенных в Кишертском районе на территории УНБ «Предуралье», и с 28 растений естественной популяции, расположенной в 500 м северо-восточнее д. Заборье Добрянского района Пермского края. Выделение ДНК проводили с использованием СТАВ-метода [15], модифицированного нами для хвойных видов [10]. Концентрацию и качество полученной ДНК определяли с помощью прибора Nanodrop ND-2000 (Thermо scientific, USA). Геномную ДНК разбавляли до концентрации 10 нг/мкл в TE-буфере. Для ПЦР использовали реакционную смесь объемом 25 мкл, содержащую 2 единицы Tag-полимеразы, 2,5 мкл стандартного 10х буфера для ПЦР; 25 пМ праймера; 2,5 мМ Mg2+; 0,25 мM dNTP, 5 мкл геномной ДНК. В качестве отрицательного (К-) контроля в реакционную смесь для проверки чистоты реактивов добавляли вместо ДНК 5 мкл деионизированной воды. Амплификацию проводили в термоциклере Gene Amp PCR System 9700 («Applied Biosystems», USA) по стандартной для ISSR-метода программе [9]. Для проверки достоверности полученных ДНК-спектров опыт повторяли не менее трех раз.
Для выявления полиморфизма ДНК лиственницы был произведен выбор информативных ISSR-праймеров в соответствии со шкалой эффективности праймеров, предложенной Р.Н. Календарем и С.В. Боронниковой: от низкой (1) до высокой (5) [8]. Каждый праймер индивидуально был анализирован в ПЦР ISSR-методом с геномной ДНК. Нами протестировано 20 ISSR-праймеров, синтезированных в ЗАО «Синтол» (г. Москва), из которых отобраны 5 наиболее информативных для дальнейшего анализа (табл. 1).
Продукты амплификации разделяли электрофорезом в 1,7 % агарозном геле в 1х ТВЕ буфере. Гели окрашивали бромистым этидием и фотографировали в проходящем ультрафиолетовом свете в системе Gel-Doc XR («Bio-Rad», США). Для определения длины фрагментов ДНК использовали маркер молекулярной массы (100 bp + 1.5 + 3 Кb DNA Ladder) (ООО «СибЭнзим-М», Москва). Определение длин фрагментов проводилось с использованием программы Quantity One в системе гель-документации Gel Doc XR («Bio-Rad»,США). Для компьютерной обработки полученные результаты были представлены в виде матрицы бинарных данных. Компьютерный анализ молекулярно-генетического полиморфизма ДНК проведен с помощью компьютерных программ POPGENE 1.31 и специализированного макроса GenAlEx6 для MS-Excel с определением: доли полиморфных локусов (P95), абсолютного числа аллелей (na), эффективного числа аллелей (ne), ожидаемой гетерозиготности (HE) [15].
Результаты исследования и их обсуждение
Протестированные 20 ISSR-праймеров содержали последовательности ди- и тринуклеотидных микросателлитных мотивов с добавлением якорного нуклеотида на 3’-конце. Таким образом, из 20 протестированных ISSR-праймеров 6 показали высокую эффективность (5), так как выявили наибольшее число четко амплифицирующихся фрагментов ДНК, четыре праймера обнаружили среднюю (4) и остальные праймеры показали невысокую эффективность (3 и ниже). Для проведения молекулярно-генетического анализа полиморфизма ДНК лиственницы были отобраны следующие праймеры: (AC)8CT, (ACC)6G, (AGC)6C, (GAC)6C, (CA)6GT (табл. 1).
Таблица 1
Эффективность ISSR-праймеров
|
Праймер |
Температура отжига (Tm, °C) |
Эффективность праймера |
Праймер |
Температура отжига (Tm, °C) |
Эффективность праймера |
||
|
1 |
(AC)8CG |
56 |
3 |
11 |
(AG)8CA |
54 |
3 |
|
2 |
(AC)8CC |
56 |
1 |
12 |
(AG)8CG |
56 |
3 |
|
3 |
(AC)8CT |
54 |
5 |
13 |
(CTC)6C |
64 |
3 |
|
4 |
(GA)8C |
53 |
2 |
14 |
(GAG)6C |
56 |
5 |
|
5 |
(ACC)6G |
62 |
5 |
15 |
(ACG)7G |
64 |
4 |
|
6 |
(AGC)6C |
64 |
5 |
16 |
(ATG)7C |
60 |
4 |
|
7 |
(AGC)6G |
64 |
4 |
17 |
(CT)8TC |
54 |
2 |
|
8 |
(AC)8T |
50 |
5 |
18 |
(CA)6GT |
56 |
5 |
|
9 |
(TG)8AA |
52 |
1 |
19 |
(GA)6GG |
46 |
4 |
|
10 |
(TG)8GC |
56 |
1 |
20 |
(GT)6GG |
46 |
3 |
Примечание. Эффективность праймеров от 1 (низкая) до 5 (высокая) определена по шкале, предложенной С.В. Боронниковой и Р.Н. Календарем (2007).
Это свидетельствует о том, что структурные элементы генома, фланкированные АСА-повторами, представлены в геноме исследованного вида с большей частотой, чем участки, фланкированные GT-повторами. Обнаружено, что каждый используемый праймер в ISSR-методе приводил к формированию специфичных спектров продуктов амплификации, причем их полиморфизм не был прямо связан с количеством выявляемых локусов. Спектры, получаемые с использованием ди- и тринуклеотидных микросателлитных мотивов в качестве праймеров, оказались в равной степени насыщены ампликонами.
ISSR-маркирование природной популяции лиственницы выявило 80 амплифицированных фрагментов ДНК, из которых 74 были полиморфными (P95 = 0,925).
Число амплифицированных фрагментов ДНК варьировалось в зависимости от праймера от 11 до 26, а их размеры – от 200 до 2190 пн. Наибольшее число локусов выявил праймер (ACC)6G. Доля полиморфных локусов природной популяции лиственницы высока и варьировалась от 0,923 до 1,000 (табл. 2).
Таблица 2
Характеристика ISSR-спектров изученных популяций лиственницы
|
Праймер |
Популяция |
Границы длин локусов спектра, п.н. |
Число ампликонов в спектре |
Число полиморфных ампликонов |
Доля полиморфных ампликонов |
|
(AC)8 CT |
искусственная |
1160–260 |
30 |
30 |
1 |
|
природная |
640–280 |
11 |
10 |
0,909 |
|
|
(ACC)6G |
искусственная |
2190–200 |
19 |
17 |
0,895 |
|
природная |
2190–230 |
26 |
26 |
1 |
|
|
(AGC)6C |
искусственная |
1190–270 |
8 |
2 |
0,250 |
|
природная |
1190–220 |
17 |
14 |
0,823 |
|
|
(GAG)6C |
искусственная |
1960–290 |
21 |
19 |
0,905 |
|
природная |
1660–240 |
11 |
10 |
0,909 |
|
|
(CА)6GT |
искусственная |
1130–200 |
32 |
28 |
0,875 |
|
природная |
620–200 |
15 |
14 |
0,933 |
Анализ ISSR-спектров лиственницы из искусственных насаждений выявил 110 амплифицированных фрагментов ДНК, из которых 96 (P95 = 0,872) были полиморфными. Число амплифицированных ISSR-фрагментов в зависимости от праймера варьировалось от 8 до 32. Доля полиморфных локусов искусственной популяции лиственницы ниже и варьировалась от 0,250 до 1,000. Размеры ISSR-фрагментов у растений из искусственных насаждений лиственницы изменялись так же как, и у растений природной популяции – от 200 до 2190 пн (рисунок). Однако ISSR-спектры искусственной популяции характеризуются наличием длинных фрагментов ДНК в отличие от таковых природной популяции, особенно четко это отражали спектры динуклеотидных праймеров (AC)8CT и (СА)6GT (табл. 2).
Праймеры (GAG)6C и (AGC)6C выявили сходные спектры по диапазону длин ампликонов, но отличаются по доле полиморфных локусов. Так, ISSR-спектры, выявляемые с использованием в ПЦР праймера (AGC)6C, оказались менее полиморфными для обеих выборок (P95 = 0,250 для искусственной, P95 = 0,823 для естественной популяции). Высокая доля полиморфных локусов установлена с использованием праймера (GAC)6C (0,905 и 0,909 соответственно) (рисунок).
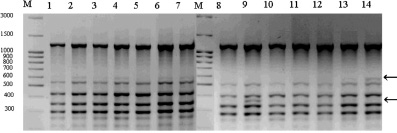
Фрагмент ISSR-спектра искусственной популяции с праймером (AGC)6C: М – маркер молекулярного веса; 1–14 – номера проб ДНК, стрелками обозначены некоторые полиморфные фрагменты
Сравнительный анализ генетических структур по полилокусным спектрам продуктов амплификации ISSR-маркеров различных по происхождению популяций лиственниц показал, что в искусственной популяции 44,5 % выявленных локусов являлись уникальными, а в природной популяции – только 25 %. Примечательно, что фрагмент длинной 220 п.н. спектра праймера (AGC)6C являлся мономорфным для природной популяции и не был отмечен нами ни у одного растения из искусственной популяции. Этот факт позволяет обозначить этот ISSR-маркер как идентификационный для природной популяции. Мономорфными для искусственной популяций являлись 7 (0,53 %) амплифицированных фрагментов ДНК, но в природной популяции они встречались с низкой частотой. В природной популяции отмечено только 3 (0,02 %) мономорфных фрагмента. Интересено, что из 10 мономорфных фрагментов ДНК, отмеченных в двух изученных популяциях, только 6 (0,46 %) встречались в обеих популяциях (табл. 3).
На основании проведенного анализа полиморфизма ДНК выполнена оценка параметров генетического разнообразия исследованных популяций (табл. 4).
Таблица 3
Частота ISSR-маркеров двух изученных популяций лиственницы
|
Праймер |
Длины фрагментов, п.н. |
Частота |
|
|
искусственная популяция |
природная популяция |
||
|
(AC)8 CT |
640 |
0,308 |
1,000 |
|
500 |
0,500 |
1,000 |
|
|
(ACC)6G |
620 |
1,000 |
0,643 |
|
410 |
1,000 |
1,000 |
|
|
(AGC)6C |
1190 |
1,000 |
0,214 |
|
540 |
1,000 |
0,643 |
|
|
440 |
1,000 |
0,678 |
|
|
350 |
1,000 |
1,000 |
|
|
300 |
1,000 |
1,000 |
|
|
270 |
1,000 |
0,143 |
|
|
220 |
0 |
1,000 |
|
|
(GAG)6C |
610 |
1,000 |
1,000 |
|
530 |
1,000 |
1,000 |
|
|
350 |
0,231 |
0,857 |
|
|
(CA)6GT |
350 |
1,000 |
0,500 |
|
300 |
1,000 |
0,714 |
|
|
200 |
1,000 |
1,000 |
|
Таблица 4
Показатели генетического разнообразия двух популяций лиственницы
|
Популяция |
HE |
na |
ne |
P95 |
|
Искусственные насаждения |
0,197 (0,014) |
1,740 (0,440) |
1,302 (0,294) |
0,872 |
|
Естественная популяция |
0,161 (0,016) |
1,557 (0,498) |
1,263 (0,335) |
0,925 |
|
На общую выборку |
0,210 (0,013) |
1,954 (0,209) |
1,329 (0,299) |
0,894 |
Примечание: HE – ожидаемая гетерозиготность; na – абсолютное число аллелей на локус; ne– ффективное число аллелей на локус; у всех вышеуказанных параметров в скобках даны стандартные отклонения.
Определенно, что природная популяция характеризуется более высокой долей полиморфных локусов (P95 = 0,925), а остальные показатели генетического разнообразия ниже у этой популяции (HE = 0,161; na = 1,557; ne = 1,263).
Заключение
Из 20 протестированных ISSR-праймеров выявлены пять эффективных для анализа полиморфизма ДНК лиственницы. Выполнен сравнительный анализ генетических структур по полилокусным спектрам продуктов амплификации ISSR-маркеров двух различных по происхождению популяций лиственниц. Анализ ISSR-спектров растений природной популяции лиственницы выявил 80 амплифицированных фрагментов ДНК, из которых 74 полиморфны (P95 = 0,925), у деревьев из искусственных насаждений – 110 амплифицированных фрагментов ДНК, из которых полиморфны 96 (P95 = 0,872). Доля полиморфных локусов природной популяции лиственницы высока и варьировалась от 0,923 до 1,000, а у искусственной популяции этот показатель изменялся от 0,250 до 1,000, то есть искусственная популяция содержит генетически более гомогенные деревья. Выявлен идентификационный уникальный ISSR-маркер для природной популяции лиственницы. В дальнейших исследованиях данный фрагмент может быть использован при составлении молекулярно-генетической формулы по предложенной С.В. Боронниковой [3] методике молекулярно-генетической идентификации и паспортизации растений, в соответствии с которой мы обозначили его как Lsv220(AGC)6C. Показано, что ISSR-спектры искусственной популяции характеризуются наличием длинных фрагментов ДНК в отличие от таковых природной популяции, особенно четко это отражали спектры динуклеотидных праймеров.
Наибольшее число локусов выявил праймер (ACC)6G. Показано, что структурные элементы генома лиственницы, фланкированные АСА-повторами, представлены с большей частотой, чем участки, фланкированные GT-повторами. Праймеры (GAG)6C и (AGC)6C выявили сходные спектры по диапазону длин ампликонов, но выраженные отличия по доле полиморфных локусов. Эти последовательности принадлежат к так называемым пурин/пиримидиновым трекам, способным формировать триплексные структуры, предположительно, принимающим участие в регуляции генной экспрессии [12]. При оценке параметров генетического разнообразия исследованных популяций лиственницы определено, что природная популяция характеризуется более высокой долей полиморфных локусов (P95 = 0,925), а остальные показатели генетического разнообразия ниже у этой популяции (HE = 0,161; na = 1,557; ne = 1,263).
Таким образом, сравнительный анализ генетических структур по полилокусным спектрам продуктов амплификации ISSR-маркеров различных по происхождению популяций лиственниц позволил установить в геноме этого вида наличие определенных микросателлитных повторов, определить основные параметры генетического разнообразия популяций и выявить идентификационные фрагменты и их сочетания для каждой популяции.
Работа выполнена в соответствии с государственным заданием на оказание услуг, частично финансируемых Министерством образования и науки РФ из средств федерального бюджета, на оборудовании, закупленном в ходе реализации проекта развития Пермского национального исследовательского университета.
Рецензенты:
Переведенцева Л.Г., д.б.н., профессор кафедры ботаники и генетики растений биологического факультета, ФГБОУ ВПО «Пермский государственный национальный исследовательский университет», г. Пермь;
Новоселова Л.В., д.б.н., профессор кафедры ботаники и генетики растений биологического факультета, ФГБОУ ВПО «Пермский государственный национальный исследовательский университет», г. Пермь.
Работа поступила в редакцию 03.06.2013.



